1. Los ácidos nucleicos
1.1 Perspectiva histórica
En los años 40 hubo un movimiento científico encaminado a intentar descubrir cuál era la naturaleza del gen. Hasta entonces este había sido tratado en la genética clásica o mendeliana, que estudiaremos el tema que viene, pero no se había indagado en su naturaleza fisicoquímica.
En 1868, un médico alemán llamado Friedich Miescher (Figura 19.3) descubrió, cuando analizaba la composición de núcleos de células del pus procedente de vendajes quirúrgicos, un cuarto tipo de sustancia que se añadía a los ya por entonces conocidos glúcidos, lípidos y proteínas como componente esencial de la materia viva. Se trataba de una sustancia ácida, rica en fósforo, que Miescher denominó nucleína y que poco después empezó a conocerse con el nombre de ácido nucleico, denominación esta que hacía referencia tanto a su carácter ácido como a su localización en el núcleo celular.
Resulta paradójico que esta sustancia fuese descubierta sólo tres años después de que Mendel estableciese el concepto de gen y que hubiesen de transcurrir otros noventa años hasta que dicha sustancia fuese reconocida universalmente como su soporte material. En ambos casos los descubrimientos permanecieron durante décadas sin recibir la atención debida.
La teoría cromosómica de la herencia ya establecía que los genes estaban localizados en los cromosomas que se encontraban en el núcleo de las células. El análisis químico de estos mostraba que estaban formados por ADN y proteínas en proporciones similares y, por lo tanto, una de las dos moléculas debía ser la responsable de contener la información genética. Curiosamente la mayoría se inclinaba por las proteínas al considerar que tenían una variabilidad mucho mayor (no se conocía cuál era la estructura del ADN).
En 1928, F. Griffith (fig19.4) estudiaba el proceso de infección en ratones por Streptococcus pneumoniae, más conocido como "neumococo", una bacteria que se encuentra entre los agentes causantes de la neumonía humana y que resulta especialmente patógena para el ratón: la inyección en un ratón de esputos procedentes de un paciente afectado de neumonía neumocócica le ocasiona a aquél la muerte en menos de 24 horas. El neumococo debe su carácter patógeno a una cápsula de polisacáridos que lo protege de los mecanismos de defensa del animal infectado. Griffith había aislado una cepa mutante de esta especie, que había perdido su capacidad para sintetizar esta cápsula y que resultaban por lo tanto vulnerables a dichos mecanismos de defensa: los ratones inoculados con bacterias de esta cepa no contraían la neumonía y por consiguiente sobrevivían. Ambas variantes podían distinguirse una de la otra con facilidad debido al aspecto de las colonias que formaban en las placas de cultivo, que tenían aspecto brillante (S) en la variante patógena común y aspecto rugoso (R) en la variante mutante no patógena. El aspecto brillante o rugoso de las colonias era también una consecuencia de la presencia o ausencia respectivamente de la mencionada cápsula de polisacáridos.
En el curso de sus investigaciones Griffith descubrió con sorpresa que los ratones inoculados con mutantes R no patógenos mezclados con una muestra de bacterias S patógenas previamente muertas por efecto del calor, contraían la neumonía y morían a las pocas horas.
Las bacterias recuperadas de la sangre de los ratones muertos habían recuperado su capacidad para sintetizar la cápsula de polisacáridos y con ello su carácter patógeno y el aspecto brillante de las colonias a las que daban lugar. El contacto con las bacterias S había producido en las bacterias R una transformación R → S que se transmitía a las sucesivas generaciones celulares.
Años más tarde Griffith comprobó que no era imprescindible que el contacto entre las dos cepas bacterianas se produjese en el interior del ratón: la transformación también se producía en cultivos de bacterias R que crecían en contacto con bacterias S muertas.
Más significativo aún resultó el hecho de que la transformación se produjese como consecuencia del contacto de cultivos de bacterias R creciendo en contacto con un "extracto libre de células" de bacterias S, es decir, no era imprescindible la estructura celular intacta de las bacterias S muertas sino que una disolución de sus componentes moleculares solubles era suficiente.
Los experimentos de Griffith fueron el punto de partida del trabajo de Avery, McLeod y McCarthy, que se plantearon identificar, en el extracto libre de células que se ha mencionado, la naturaleza química del "principio transformante" responsable del fenómeno observado.
Para ello llevaron a cabo un fraccionamiento sistemático del extracto libre de células y ensayaron la capacidad transformante de las distintas fracciones sobre cultivos de bacterias R. Tras ensayar con distintas fracciones del extracto (lípidos, glúcidos, proteínas, etc.) con resultados negativos, comprobaron que la fracción que contenía los ácidos nucleicos inducía eficazmente la transformación. Un fraccionamiento ulterior llevó a la conclusión de que el principio transformante buscado no era otro que el DNA bacteriano: pequeñísimas cantidades de este DNA purificado eran suficientes para transformar las bacterias R en bacterias S. Avery y sus colaboradores demostraron también que el DNA de las bacterias transformadas y de sus descendientes podía a su vez inducir la transformación en otras bacterias R y que en sucesivos ciclos de transformación como los descritos se mantenía esta capacidad.
Tras estas experiencias las conclusiones de Avery (Figura 19.6) estaban cada vez más claras: el DNA de las bacterias S muertas era la sustancia que contenía la información necesaria para hacer que las bacterias R y su descendencia recuperasen su capacidad para sintetizar su cápsula de polisacáridos y con ella su carácter patógeno, es decir, el DNA era el material genético de Streptococcus pneumoniae.
Muchos autores seguían creyendo que eran las proteínas las que tenían que tener la información genética y El equipo de Avery trató el extracto el DNA purificado de las bacterias S con proteasas (enzimas que degradan las proteínas) sin que esto afectara a su actividad transformante. Por otra parte, el tratamiento con desoxirribonucleasas (enzimas que degradan el DNA) destruía en pocos minutos cualquier rastro de dicha actividad. Otros experimentos, realizados por R.D. Hotchkiss, confirmaron que la actividad transformante del DNA no se restringía al carácter virulento o no de las cepas bacterianas, ni al aspecto liso o rugoso de sus colonias, sino que también operaba de manera análoga para otros caracteres hereditarios, como la resistencia a distintos tipos de antibióticos.
La principal dificultad para aceptar al ADN como molécula hereditaria es que solo se sabía que estaba formada por cuatro nucleótidos (dAMP, dGMP, dTMP y dCMP) y se pensaba que como con los glúcidos era una molécula con repeticiones monótonas y que no podía codificar información (incluso cuando se descubrió que su tamaño era MUY superior al esperado).
El trabajo de Chagaff (fig 19.7), con análisis mucho más precisos de la composición de nucleótidos del ADN, permitió establecer que la proporción de los mismos variaba lo que acababa con la idea de molécula monótona y permitía alumbrar la idea de que estas variaciones codificaran información y, por lo tanto, que el ADN fuera la molécula que contenía la información genética. Un descubrimiento adicional, enunciado bajo la regla de equivalencia de Chagaff, revela que el número de bases púricas es igual al de pirimidínicas y que el número de A es igual al número de T y el número de G es igual al número de C.
En 1952, Chase y Hershey (Figura 19.8) diseñaron un experimento con el objeto de elucidar los detalles del proceso de infección de células bacterianas de la especie Escherichia coli por bacteriófagos T4.
Las partículas infecciosas de este fago están compuestas exclusivamente por DNA y proteínas y Hershey y Chase querían saber como se comportaba uno y otro tipo de macromoléculas durante el proceso de infección. Para ello, idearon una ingeniosa técnica de marcaje mediante isótopos radiactivos. Se percataron de que en las partículas virales la práctica totalidad de los átomos de fósforo se encontraban en el DNA (en los grupos fosfato de la cadena polinucleotídica) mientras que la práctica totalidad de los átomos de azufre se encontraban en las proteínas (en los aminoácidos metionina y cisteína). Así, decidieron utilizar los isótopos radiactivos 32P y 35S para delatar respectivamente la presencia de DNA y de proteínas.
Para obtener partículas víricas marcadas permitieron el crecimiento de un cultivo de fagos T4 sobre células de E. coli en un medio en el que la única fuente de fósforo eran iones fosfato (PO43-) marcado con 32P, de manera que este isótopo se incorporaba a todas las biomoléculas de las células bacterianas y de los fagos que se reproducían en su interior.
Por otra parte, hicieron lo propio con fagos obtenidos en un cultivo con iones sulfato (SO42-) marcado con 35S, que se incorporaría igualmente a las biomoléculas de bacterias y fagos.
De este modo, una vez aislados y purificados los fagos obtenidos en uno y otro cultivo, dispusieron de dos cepas virales, una de ellas con el DNA marcado con 32P y otra con las proteínas marcadas con 35S. (Figura 19.8b)
Posteriormente se infectó a dos cultivos de E. coli con cada una de las cepas virales y, tras esperar un tiempo y separar los componentes por centrifugación, los resultados mostraban claramente que de los dos componentes de la partícula vírica sólo el DNA penetraba en el interior de la célula durante el proceso de infección.
Las proteínas del fago permanecían en el exterior de la célula durante todo el proceso de infección y se desprendían de la superficie celular por agitación.
Es decir, la partícula vírica infecciosa se fija a la superficie celular y de alguna manera “inyecta” su DNA en el interior de la célula. La cápside proteica, una vez inyectado en la bacteria el DNA que albergaba en su interior, ya no resulta más necesaria en el proceso de infección, como prueba el hecho de que la eliminación de estos “fagos fantasma” por agitación no altere la capacidad de las células infectadas para dar lugar a nuevas progenies virales.
Es la molécula de DNA vírico la que, una vez dentro de la célula, parece tomar el control de su metabolismo para que éste se ponga al servicio del virus y comience a fabricar nuevas partículas infecciosas atendiendo a las instrucciones cifradas en esa misma molécula. En otras palabras: el DNA es el material genético del bacteriófago T4.
Ya solo quedaba averiguar cuál es la estructura real del ADN pero eso lo veremos después de estudiar brevemente sus componentes.
1.2 Nucleótidos
Al igual que todas las biomoléculas orgánicas, los ácidos nucleicos son polímeros formados por la unión de unidades de menor tamaño o monómeros: los nucleótidos.
Al contrario que en otras biomoléculas, los nucleótidos están a su vez formados por tres moléculas más sencillas: una pentosa, una molécula de ácido fosfórico y una base nitrogenada enlazados de un modo característico.
Las pentosas que aparecen formando parte de los nucleótidos son la β-D-ribosa y su derivado, el desoxiazúcar 2'-β-D-desoxirribosa, en el que el grupo hidroxilo unido al carbono 2' fue sustituido por un átomo de hidrógeno.
El tipo de ácido fosfórico que se encuentra en los nucleótidos es concretamente el ácido ortofosfórico.
Las bases nitrogenadas son compuestos heterocíclicos que, gracias al sistema de dobles enlaces conjugados que poseen en sus anillos, poseen un acusado carácter aromático, siendo su conformación espacial planar o casi planar. Sus átomos de nitrógeno poseen pares electrónicos no compartidos que tienen tendencia a captar protones, lo que explica su carácter débilmente básico. Los compuestos originarios de los que derivan estas bases nitrogenadas son la purina y la pirimidina.
Las pentosas se unen a las bases nitrogenadas dando lugar a unos compuestos denominados nucleósidos.
Los nucleótidos resultan de la unión mediante enlace éster de la pentosa de un nucleósido con una molécula de ácido fosfórico. Esta unión, en la que se libera una molécula de agua, puede producirse en cualquiera de los grupos hidroxilo libres de la pentosa, pero como regla general tiene lugar en el que ocupa la posición 5'; es decir, los nucleótidos son los 5' fosfatos de los correspondientes nucleósidos. La posesión de un grupo fosfato, que a pH 7 se encuentra ionizado, confiere a los nucleótidos un carácter marcadamente ácido.
Al igual que los nucleósidos, los nucleótidos pueden clasificarse en ribonucleótidos y desoxirribonucleótidos según contengan ribosa o desoxirribosa respectivamente. Existen diversas maneras de nombrar los nucleótidos; cada nucleótido se identifica mediante tres letras mayúsculas, la primera de ellas es la inicial de la base nitrogenada, la segunda indica si el nucleótido es Mono~, Di~, o Trifosfato, y la tercera es la inicial del grupo fosfato; en el caso de los desoxirribonucleótidos se antepone una "d" minúscula a estas tres siglas.
1.3. Los ácidos nucleicos: enlace o puente fosfodiester
Los ácidos nucleicos son polímeros de nucleótidos. En ellos la unión entre las sucesivas unidades nucleotídicas se realiza mediante enlaces tipo éster-fosfato que resultan de la reacción entre el ácido fosfórico unido al carbono 5' de la pentosa de un nucleótido y el hidroxilo del carbono 3' de la pentosa de otro nucleótido. Este tipo de unión, en la que un grupo fosfato queda unido por dos enlaces éster a dos nucleótidos sucesivos, se conoce también como puente fosfodiéster.
Cuando dos nucleótidos se unen mediante un puente fosfodiéster el dinucleótido que resulta conserva un grupo 5' fosfato libre en un extremo que puede reaccionar con el grupo hidroxilo 3' de otro nucleótido, y un grupo hidroxilo 3' libre que puede reaccionar con el grupo 5' fosfato de otro nucleótido. Esta circunstancia permite que mediante enlaces fosfodiéster se puedan enlazar un número elevado de nucleótidos para formar largas cadenas lineales que siempre tendrán en un extremo un grupo 5' fosfato libre y en el otro un grupo hidroxilo 3' libre.
1.4. El ácido desoxirribonucleico o ADN
El ADN es un ácido nucleico constituido por la polimerización de desoxirribonucleótidos. Es una molécula formada por dos cadenas polinucleótidas antiparalelas enrolladas en una doble hélice. Las dos cadenas permanecen unidas gracias a los enlaces de puentes de hidrógeno que se establecen entre las bases nitrogenadas de ambas.
Normalmente se encuentra en el núcleo de la célula (ADN nuclear), en el cromosoma bacteriano, en el interior de las mitocondrias (ADN mitocondrial) o de los cloroplastos (ADN plastidial).
Las principales funciones del ADN son:
a) El ADN contiene la información genética codificada en la secuencia de sus bases nitrogenadas. Según sean su orden y su distribución, así serán las proteínas que se sinteticen siguiendo las instrucciones del código genético, que hacen corresponder la secuencia de bases nitrogenadas del ADN con la secuencia de aminoácidos de la proteína.
b) La segunda propiedad del ADN es que puede duplicarse (replicación) lo que asegura la conservación de la información genética y su transmisión a la descendencia.
1.4.1. Estructura 1ª del ADN
La estructura primaria del ADN es la secuencia de nucleótidos (unidos por enlaces fosfodiéster) de una sola cadena o hebra, que puede presentarse como un simple filamento extendido o bien algo doblada en sí misma.
Como hemos visto, una cadena de ADN presenta dos extremos libres: el 5´, unido al grupo fosfato, y el 3´, unido a un hidroxilo.
A comienzos de los años 50, tres centros de investigación rivalizaban en el análisis de la estructura tridimensional de las biomoléculas mediante cristalografía de rayos X. Uno de ellos era el Instituto de Tecnología de California, cuya división de química, dirigida por Linus Pauling, se había apuntado varios éxitos notables en el descubrimiento de la estructura secundaria de las proteínas.
Otro era el Laboratorio Cavendish de la Universidad de Cambridge (Inglaterra). Fue en el Laboratorio Cavendish donde coincidieron a comienzos de 1951 dos jóvenes investigadores, James D. Watson y Francis H.C. Crick, que estaban llamados a ser quienes desvelaran finalmente la estructura de la molécula de ADN.
Un tercer grupo se había formado en el King’s College de Londres bajo la jefatura de John Randall, que contaba con la colaboración de Maurice Wilkins y de la experta cristalógrafa Rosalind Franklin.
Entre 1951 y 1953 se desató entre estos grupos una especie de carrera por identificar la estructura tridimensional del ADN, carrera que se aceleró cuando la publicación del experimento de Hershey y Chase a finales de 1952 puso a todos los grupos sobre la pista correcta de cual era en realidad la molécula de la herencia.
En el King’s College, Rosalind Franklin había desarrollado una técnica que le permitía obtener fibras de ADN altamente orientadas y obtener así difractogramas de rayos X de una calidad y lujo de detalles muy superiores a los conocidos hasta entonces.
Mientras tanto Watson y Crick trataban de construir su propio modelo tridimensional basándose en difractogramas de una calidad muy inferior. No obstante, su trabajo se encontraba muy avanzado; habían analizado cuidadosamente la estructura de los nucleótidos individuales y se habían percatado de que los datos obtenidos por Chargaff tres años antes, sobre las proporciones de las bases nitrogenadas (comprobó que todos los ADN tenían el mismo número de A que de T y de C que de G), debían tener algún significado relevante, lo que probablemente fue una de las claves de su éxito posterior.
Fue entonces cuando Jim Watson, durante una conversación con Maurice Wilkins, pudo ver algunos de los difractogramas obtenidos por Rosalind Franklin; la simple inspección visual de aquellos difractogramas proporcionó a Watson las claves que le faltaban para resolver finalmente la estructura del ADN.
En pocas semanas Watson y Crick terminaron de encajar sus propios datos con lo que se apreciaba en los difractogramas de Franklin y elaboraron un modelo definitivo que fue publicado en el número de abril de la revista Nature. La carrera había terminado.
El modelo propuesto por Watson y Crick, mundialmente conocido como la doble hélice, presentaba las siguientes características:
a) La molécula de ADN está formada por dos cadenas polinucleotídicas antiparalelas, es decir, si una cadena se recorre en dirección 5’—›3’, su vecina discurriría en dirección 3’—›5’.
b) Ambas cadenas se encuentran formando un arrollamiento helicoidal de tipo plectonémico, es decir, para separarlas habría que desenrollarlas girando una sobre la otra. El arrollamiento es además dextrógiro.
c) El conjunto forma una estructura cilíndrica con un diámetro constante de 2 nm.
d) Los esqueletos azúcar-fosfato de las cadenas polinucleotídicas se encuentran en el exterior de la estructura, formando lo que serían las guías de una especie de escalera de caracol.
e) Las bases nitrogenadas se proyectan desde los esqueletos azúcar-fosfato hacia el interior de la estructura y se disponen apiladas por pares formando lo que equivaldría a los peldaños de la escalera.
f) Los pares de bases nitrogenadas están formados invariablemente por una purina y una pirimidina (complementarias). Además, siempre se encuentran enfrentadas adenina con timina por una parte y guanina con citosina por otra.
g) Las dos cadenas polinucleotídicas se encuentran unidas por puentes de hidrógeno entre grupos funcionales de las bases nitrogenadas que forman cada par. Cada adenina forma dos puentes de hidrógeno con la correspondiente timina y cada guanina tres con la citosina. Pares de bases diferentes a los establecidos no podrían formar puentes de hidrógeno.
h) La distancia entre cada par de bases sucesivo es de 0,34 nm. Cada vuelta completa de la hélice (paso de rosca) alberga exactamente 10 pares de nucleótidos, lo que se corresponde con una longitud de 3,4 nm. Ambas periodicidades aparecían reflejadas en los difractogramas.
Uno de los aspectos más interesantes del modelo de Watson y Crick residía en que no sólo encajaba con los datos de difracción de rayos X sino que además proporcionaba una explicación para la hasta entonces desconcertante regla de Chargaff. En efecto, si todos los pares de bases eran necesariamente A-T o G-C, en cualquier muestra de DNA el número de restos de adenina debía ser igual al de timina y el de guanina al de citosina, de lo que se deduce que el número total de bases púricas debería ser igual al de bases pirimídicas.
Además, este emparejamiento específico de las bases nitrogenadas podría encerrar un profundo significado biológico, pues, como Watson y Crick sugerían en su artículo en Nature, tal emparejamiento podría ser la base del mecanismo por el que el material genético creaba copias de si mismo en cada ciclo de reproducción celular. La complementariedad interna de la doble hélice, regida por la regla de Chargaff, hacía que cada una de las dos cadenas polinucleotídicas que la formaban pudiera ser utilizada como molde para sintetizar otra con una secuencia de bases complementaria.
La publicación del modelo de Watson y Crick en abril de 1953 y la gran difusión que tuvo en los meses posteriores diluyó rápidamente cualquier resto de escepticismo acerca del papel del ADN como material hereditario, que ya no volvió a ser discutido. Todo ello supuso una auténtica revolución en el seno de las ciencias biológicas y el nacimiento de lo que se dio en llamar biología molecular, área del conocimiento que tuvo un gran desarrollo en las décadas siguientes y que ha contribuido de forma decisiva a nuestra comprensión actual del funcionamiento de los sistemas vivos.
Como ya vimos el tema pasado, el ADN tiene una estructura terciaria y cuaternaria derivada de su asociación con proteínas dando lugar a la cromatina y los cromosomas (repasar el tema anterior).
1.5. El ácido ribonucleico o ARN
Se comprobó que en las células eucariotas la casi totalidad del ADN celular se encuentra en el interior del núcleo mientras que la mayor parte del ARN se encuentra en el citoplasma (aunque algunas zonas del núcleo, en particular el nucléolo, también son ricas en ARN). Por otra parte, del total de ARN citoplasmático una fracción muy importante se encontraba asociado a determinadas proteínas para formar unas partículas, visibles al microscopio electrónico, que fueron denominadas ribosomas. Experimentos realizados utilizando aminoácidos marcados radiactivamente pronto demostraron que los ribosomas eran el lugar de la célula donde se llevaba a cabo la síntesis de las proteínas, por lo que ya desde entonces se asoció al ARN con este proceso.
La estructura tridimensional del ARN difiere claramente de la del ADN. En general las moléculas de ARNson monocatenarias (una sola cadena polinucleotídica). Sin embargo, existen moléculas de ARN que, aun siendo monocatenarias, presentan tramos con secuencias de bases complementarias los cuales adoptan estructuras en doble hélice, denominadas horquillas, de características análogas a las del ADN. En ocasiones, cuando las secuencias complementarias no son contiguas, se forman bucles de bases no emparejadas dentro de las horquillas. En las dobles hélices de ARN la adenina se empareja con el uracilo, que tiene estructura similar e idénticas posibilidades de formar puentes de hidrógeno que la timina, y la guanina con la citosina.
Hoy sabemos que la función primordial del ARN en las células consiste en servir de intermediario para transferir la información genética cifrada en el ADN a la estructura tridimensional de las proteínas en el proceso de expresión de la información genética.
De todos modos, en algunos virus el ARN constituye en sí mismo el material genético además de servir de intermediario en el proceso de síntesis de las proteínas virales. En algunos de estos virus la molécula de ARN que constituye el cromosoma viral es bicatenaria y presenta en toda su longitud estructura de doble hélice. También existen virus con cromosomas de ARN monocatenario.
Existen varios tipos de ARN que difieren en el tamaño, estructura y función específica de sus moléculas. Todos ellos se sintetizan en el núcleo celular a partir de secuencias de ADN que sirven como molde y una vez sintetizados atraviesan los poros nucleares y se incorporan a sus diferentes destinos en el citoplasma:
1.5.1 ARN ribosómico (ARNr)
El ácido ribonucleico ribosómico o ribosomal (ARNr) es el tipo de ARN más abundante (80-85% del ARN total) en las células y constituye, en un 60% del peso, los ribosomas. Estos se encargan de la síntesis de proteínas según la secuencia de nucleótidos presente en el ARN mensajero.
Las células procariotas presentan ribosomas de 70 S, menor peso que los de las células eucariotas, de 80 S.
Sus moléculas son relativamente pequeñas (75 a 90 nucleótidos de longitud). Presentan una estructura característica con horquillas y bucles que les dan el aspecto de hojas de trébol cuando se representan sobre un plano: su estructura tridimensional presenta en realidad forma de L invertida. El ARNt presenta bases nitrogenadas diferentes a las características de los ácidos nucleicos en una proporción que puede alcanzar el 10% del total. Su función consiste en transportar de manera específica a los diferentes aminoácidos hasta los ribosomas para que allí sean ensamblados en las cadenas polipeptídicas en formación. Existen alrededor de 50 tipos de ARNts que difieren en sus secuencias de nucleótidos y en algunos aspectos de su conformación tridimensional; sin embargo todos ellos comparten algunas características:
· en el extremo 5’ de la cadena polinucleotídica hay un triplete de bases nitrogenadas una de las cuales es siempre guanina.
· en el extremo 3’ la cadena polinucleotídica finaliza con la secuencia CCA y estas bases no están emparejadas. En este lugar es donde el ARNt se une a su aminoácido correspondiente.
· la molécula presenta tres brazos cada uno de los cuales consta de una horquilla con estructura en doble hélice y un bucle formado por bases sin emparejar (Figura 19.16). Se distinguen el brazo T (por donde la molécula se une al ribosoma), el brazo D (lugar que reconocen los enzimas específicos que unen los ARNt con sus aminoácidos correspondientes) y el brazo A (cuyo bucle presenta un triplete de bases, denominado anticodón, que es complementario de otro triplete, llamado codón, que se encuentra en el ARN mensajero, siendo esta complementariedad de gran importancia en el proceso de síntesis de proteínas).
El ARNm es monocatenario, básicamente lineal y sólo constituye el 2-5 % del ARN total.
La función del ARNm es tomar la información del ADN, que está en núcleo, y llevarla al citoplasma, donde están los ribosomas en los que se sintetizarán las proteínas con los aminoácidos aportados por los ARNt.
El ARNm se forma a partir de una hebra del ADN en un proceso llamado transcripción. Se crea, con las bases nitrogenadas complementarias, un molde con la información genética necesaria para la síntesis de proteínas. El tamaño del ARNm depende del tamaño de la proteína para la que lleva información.
En el citoplasma, las enzimas ribonucleasas, lo van a ir destruyendo para evitar la producción innecesaria de proteínas. Cuando se vuelva a necesitar la síntesis de una proteína concreta, se creará nuevo ARNm.
La información que contiene el ARNm se presenta en una secuencia de bases nitrogenadas, agrupadas en tripletes o codones, cada uno de los cuales determina la unión de un determinado aminoácido.
El ARNm tiene distinta estructura en procariotas y en eucariotas:
A) ARNm en eucariotas; el ARN mensajero obtenido después de la transcripción se conoce como ARN transcrito primario o ARN precursor o pre-ARN, que en la mayoría de los casos no se libera del complejo de transcripción en forma totalmente activa, sino que ha de sufrir modificaciones antes de ejercer su función (procesamiento o maduración del ARN). Entre esas modificaciones se encuentran la eliminación de fragmentos (splicing), la adición de otros no codificados en el ADN y la modificación covalente de ciertas bases nitrogenadas.
El ARN transcrito primario (pre-ARNm), está formado por dos tipos de segmentos que se alternan:
· Exones, segmentos con información.
· Intrones, segmentos sin información que serán suprimidos y no aparecen en el ARNm.
El ARNm eucariótico posee en su extremo 5' una caperuza (CAP), formada por un nucleótido derivado de la guanina, que da estabilidad al ARNm y permite el acceso al ribosoma para la síntesis de proteínas. Después hay un segmento sin información, seguido de otro segmento con información que empieza con las bases AUG.
En el extremo 3' aparecen unos 200 nucleótidos de adenina, la llamada “cola” de poli-A, que protege la molécula frente a las enzimas exonucleasas.
El ARNm eucariótico es monocistrónico, es decir, sólo contiene información para sintetizar una cadena polipeptídica.
B) ARNm en procariotas; el proceso de transcripción y el de traducción se realizan de manera similar que en las células eucariotas. La diferencia fundamental está en que, en las procariotas, el ARN mensajero no pasa por un proceso de maduración y, por lo tanto, no se le añade caperuza ni cola ni se le quitan intrones. Además, no tiene que salir del núcleo como en las eucariotas, porque en las células procariotas no hay un núcleo definido.
El ARNm en procariotas es policistrónico, contiene informaciones separadas para sintetizar distintas proteínas.
1.5.4 ARN nucleolar (ARNn)
El ARNn forma parte del nucléolo. Se origina a partir de la región del ADN denominada región organizadora nucleolar (NOR). Este ARN monocatenario de 45 S se asocia a proteínas procedentes el citoplasma para formar las subunidades de los ribosomas.
2. Replicación del ADN
2.1 Perspectiva histórica
Había tres posibles opciones para explicar la replicación del ADN:
a) Modelo conservativo; proponía que tras la replicación se mantenía la molécula original de DNA intacta, obteniéndose una molécula idéntica de DNA completamente nueva, es decir, con las dos hebras nuevas.
b) Modelo semiconservativo; se obtienen dos moléculas de DNA hijas, formadas ambas por una hebra original y una hebra nueva.
c) Modelo dispersivo; el resultado final son dos moléculas nuevas formadas por hebras en las que se mezclan fragmentos originales con fragmentos nuevos. Todo ello mezclado al azar, es decir, no se conservan hebras originales ni se fabrican hebras nuevas, sino que aparecen ambas mezcladas.
En principio el modelo de doble cadena de Watson y Crick hace pensar que el modelo semiconservativo es el más viable y estó quedó demostrado por un brillante experimento realizado por Meselson y Stahl.
En este experimento se cultivaron bacterias E. coli en un medio con nitrógeno pesado (15N) el tiempo suficiente para que todo su ADN estuviera formado por este nitrógeno.
Después se ponen estás células en un medio con nitrógeno normal (14N). Si la replicación del ADN siguiera el modelo conservativo despues de UNA división tendríamos cadenas de ADN con 15N (originales) y cadenas con 14N (copias):
Pero lo que se obtiene al hacer el experimento es:
Por lo tanto el modelo conservativo es falso. Podríamos creer todavía en el modelo dispersivo pero si este fuera cierto las células “nietas” (en la SEGUNDA división) saldrían otra vez con UNA sola banda y, sin embargo, encontramos DOS bandas:
Por lo tanto, el modelo dispersivo también es falso y el correcto es el modelo semiconservativo.
2.2 Proceso de la replicación del ADN
Como ya hemos visto, la replicación del ADN es semiconservativa. Aunque son parecidas la replicación del ADN no es igual en las células procariotas y en las eucariotas. Sin embargo este curso vamos a explicar un modelo general simplificado (los que elijáis Biología en 2º de bachillerato o en la Universidad/FPGS ya estudiaréis ambos tipos con más detalle).
La replicación del ADN puede resumirse en TRES fases:
A) FASE DE INICIACIÓN
La fase de iniciación empieza en una secuencia especial de nucleótidos llamada ORI (origen de replicación). En el caso de los eucariotas hay varios ORI en cada cromosoma que va a replicarse en varias partes a la vez (necesario pues su replicación es MUCHO más lenta que la de los procariotas).
Una enzima llamada Helicasa va a separar la doble hélice mientras otras (topoisomerasas y girasas) estabilizan la estructura. Unas proteínas estabilizadoras (SSBP) van a mantener la separación permitiendo que se forme la horquilla de replicación.
La enzima encargada de copiar la cadena de ADN y formar una complementaria nueva se llama ADN polimerasa III pero tiene DOS características especiales que condicionan este proceso:
a) no es capaz de empezar a copiar desde 0 (necesita “engancharse” a una cadena previa).
b) solo puede fabricar la nueva cadena en sentido 5' → 3' (no en sentido contrario).
Para solucionar el PRIMER problema hay una enzima llamada primasa que va a empezar la replicación del ADN formando una pequeña cadena de ARN complementario llamado primer o cebador al cuál se va a “enganchar” la ADN polimerasa III para ir fabricando la cadena complementaria de ADN. Posteriormente ese ARN será eliminado y sustituido por ADN por la ADN polimerasa I.
En la fase de elongación (alargamiento), la ADN polimerasa III va a fabricar la cadena complementaria de ADN. Sin embargo aquí nos encontramos con el segundo de los problemas que vimos antes.
En una de las dos hebras (hebra conductora o líder) no hay ningún problema porque la apertura de la doble cadena permite que la ADN pol III vaya fabricando la cadena complementaria de ADN sin ningún problema ni interrupciones.
Pero la doble cadena de ADN es antiparalela (como estudiamos antes) y, por lo tanto, en la otra hebra (hebra retardada) es imposible que esto ocurra porque la doble cadena se está abriendo por donde debería haber empezado a copiar la ADN pol III.
Este problema se soluciona copiando esa cadena a “trozos”. Estos fragmentos y el mecanismo mediante el cuál se unen formando una sola hebra fueron descubiertos por el matrimonio de científicos japoneses Tsuneko y Reiji Okazaki (ella recibió posteriormente el premio nobel). Por eso se denomina a esos “trozos” de cadena complementaria de ADN: fragmentos de Okazaki.
Por lo tanto en la hebra retardada la primasa va a ir fabricando varios primer según va quedando el trozo de la cadena de ADN molde y la ADN pol III se engancha a ellos para formar los fragmentos de Okazaki. Posteriormente la ADN pol I irá repasando la cadena y sustituyendo los primer de ARN por ADN.
Para finalizar el proceso, una enzima llamada ligasa unirá todos los fragmentos de Okazaki formando una sola cadena complementaria de ADN.
C) FASE DE TERMINACIÓN
Al final las dos horquillas de replicación (o las varias si hablamos de eucariotas) se encontrarán al haber terminado de copiar y sintetizar las dos nuevas hebras de ADN (la continua y la retardada).
Cuando esto ocurre, la ADN pol I elimina el último cebador y los fragmentos que queden serán unidos por la ligasa: ya tenemos dos dobles cadenas de ADN idénticas a la original y formadas por una de las hebras originales (hebra parental) y una hebra complementaria copiada.
La ADN pol I y III van a repasar las dobles cadenas para corregir cualquier posible error que se haya cometido en la replicación del ADN.
3. Expresión génica
Se llama expresión génica al proceso por el cual los organismos transforman en proteínas la información contenida en los ácidos nucleicos. Se lleva a cabo mediante una serie de mecanismos similares en todos los seres vivos.
3.1. Concepto de gen
En la genética clásica o mendeliana, que estudiaremos en el siguiente tema, un gen es una unidad discreta de función que se distribuye de forma ordenada en los cromosomas y se transmite de los progenitores a la descendencia.
En la genética molecular el concepto de gen se asocia estrechamente a una parte de la cadena de ADN con una secuencia concreta de bases nitrogenadas y que codifica la información para la síntesis de una proteína.
3.2. “Dogma” de la biología molecular
El llamado “dogma” central de la biología molecular fue enunciado en 1958 por Crick y resumía el funcionamiento de la biología molecular:
El ADN se autoduplicaria mediante la replicación, estudiada en el punto anterior, cuando la célula va a dividirse. Cuando fuera necesario producir una proteína, el gen del ADN que codifica la información de esa proteína se transcribe a ARNm; este sale del núcleo al citoplasma por los poros nucleares y se une a los ribosomas, en los que se produce la traducción del ARNm y la síntesis de la proteína.
a) En primer lugar se rechazó el término “dogma” por ser incompatible con la ciencia (algo que reconoció el propio Crick) por significar que es una creencia que no puede cuestionarse.
b) La segunda modificación vino con el descubrimiento de un tipo de virus, los retrovirus (como el VIH), cuyo material genético es el ARN y que tienen una enzima (la transcriptasa inversa) que es capaz de copiar su ARN → ADN.
c) La tercera modificación vino con el descubrimiento de unas proteínas, los priones, que eran capaces de multiplicarse al provocar el cambio en otras proteínas preexistentes. Son conocidas entre el gran público por causar el mal de las “vacas locas” o enfermedad de Creutzfeld-Jakob.
d) La cuarta modificación se produjo con el descubrimiento de las ribozimas. Estas son moléculas de ARN capaces de autoduplicarse sin la ayuda de proteínas (algo que hace pensar en que pudieron ser las primeras moléculas genéticas en el mismo origen de la vida).
La transcripción del ADN es el primer proceso de la expresión génica; en la transcripción genética, las secuencias de ADN son transcritas a ARN mediante una enzima llamada ARN polimerasa que sintetiza un ARN mensajero (ARNm) que mantiene la información de la secuencia del ADN. De esta manera, la transcripción del ADN también podría llamarse síntesis del ARN mensajero.
En este proceso intervienen:
a) Una cadena de ADN que actúa como molde. Sólo una de las dos cadenas de nucleótidos del ADN se transcribe (cadena molde) mientras que la otra (cadena informativa o codificante) no se transcribe. La cadena informativa o codificante tiene la MISMA secuencia de bases nitrogenadas que el ARNm formado (salvo porque la T se sustituye por U, claro).
b) Ribonucleótidos trifosfato de A, C, G y U (ATP, GTP, CTP y UTP). Se unen por un enlace éster entre el ácido fosfórico del carbono 5´ de un ribonucleótido trifosfato, y el grupo –OH, situado en posición 3´ del último ribonucleótido que se ha unido a la cadena de ARN que se está formando.
c) Enzimas. El proceso está catalizado por las ARN-polimerasas. La ARN polimerasa va a comenzar a actuar en unas zonas concretas del ADN llamadas promotor.
La transcripción en las células procariotas tiene algunas diferencias con la que realizan las células eucariotas, pero en ambas se realiza en cuatro fases sucesivas: iniciación, elongación, terminación y maduración.
A) FASE DE INICIACIÓN
El ADN tiene una región promotora con unas secuencias de bases de consenso (distintas en procariotas y eucariotas) que indican donde debe comentar la síntesis de ARN y qué cadena de ADN tiene que ser transcrita.
Después, la configuración de la ARN polimerasa abre y desenrolla una vuelta de la hélice de la molécula de ADN, creando la burbuja de transcripción, y permitiendo la síntesis de ARN a partir de la cadena que utiliza como molde.
B) FASE DE ELONGACIÓN
Después de que la ARN polimerasa haya desenrollado una vuelta de hélice del ADN, se desplazará por la cadena molde de ADN en sentido 3'→5', mientras añade ribonucleótidos a la nueva cadena de ARNm en sentido 5'→3', siendo ambas cadenas antiparalelas.
Según se va desplazando la ARN polimerasa sobre la cadena de ADN, ésta recupera su configuración inicial de doble hélice.
La ARN polimerasa selecciona el ribonucleótido trifosfato cuya base nitrogenada es complementaria al desoxirribonucleótido de la cadena de ADN molde, y lo une mediante enlace éster, desprendiéndose un grupo pirofostato (PPi). C, G, A y U del ARN se emparejan con G, C, T y A del ADN.
En los eucariotas, después de haber unido los primeros 30 nucleótidos transcritos, en el extremo 5' se añade una caperuza constituida por una metilguanosina trifosfato, que servirá como señal de inicio en el proceso de traducción.
Un ejemplo de transcripción sería:
Secuencia de ADN: 3'... ATGCGCTAG ... 5'
Secuencia de ARNm: 5'... UACGCGAUC ... 3'
C) FASE DE TERMINACIÓN
La ARN polimerasa sigue añadiendo nucleótidos hasta que llega a una zona del ADN, llamada señal de terminación. Esta zona se caracteriza por tener una secuencia palindrómica (secuencias que se leen de la misma forma de izquierda a derecha que de derecha a izquierda) con muchos nucleótidos con G y C, seguidos de varios con T. Esto permite que se autocomplete el extremo de ARN y se genere un bucle que provoca su separación del ADN. En ese momento, la ARN polimerasa se separa y se vuelve a formar la doble hélice en el ADN.
En eucariotas, el ARNm se sigue sintetizando hasta que la ARN-polimerasa II encuentra la secuencia TTATTT, que es la señal de corte que indica que termina la síntesis de ARN. Se separa el ARN, y una enzima añade al extremo final 3' una secuencia de unos 200 ribonucleótidos de adenina, la llamada cola de poli-A.
D) FASE DE MADURACIÓN
En procariotas, el ARNm recién formado ya puede utilizarse para sintetizar proteínas en el proceso de traducción. En cambio, el ARN que se ha transcrito (ARN transcrito primario) que originará los ARNt y ARNr tienen que tener un proceso de maduración antes de ser funcionales. Se cortan y unen fragmentos hasta obtener el ARN definitivo.
En la transcripción también se producen errores (uno por cada 104-105 nucleótidos unidos), más que durante la replicación del ADN, pero no son tan importantes porque no se transmiten a la descendencia.
En eucariotas, la maduración del ARN se realiza en el núcleo. Como ya se ha comentado, los precursores del ARNm, ARNr, y ARNt, no son funcionales y tienen que tener un proceso de maduración. Cuando se ha terminado este proceso, el ARNm ya puede salir del núcleo a través de los poros nucleares y llegar al citoplasma, donde realizará su función.
Los intrones, secuencias no codificantes se eliminan en un proceso de corte y empalme (splicing) en el que los exones se unen para formar la secuencia final del ARNm.
El ADN contiene la información de las proteínas que se tienen que sintetizar, pero esa información tiene que ser transcrita al ARNm para que llegue a los ribosomas que están en el citoplasma.
El ARN está escrito en un "lenguaje de nucleótidos", de sólo cuatro bases nitrogenadas (A, U, C, G), y las proteínas necesitan un "lenguaje de 20 aminoácidos". El paso de un lenguaje a otro se realiza gracias a un código, el código genético, en el que se relaciona la secuencia de nucleótidos del ARNm y la secuencia de aminoácidos de la proteína. El código genético determina qué aminoácido le corresponde a tres nucleótidos de ARNm (cada tres nucleótidos sucesivos del ARNm forma un triplete o codón que equivalen a un aminoácido de la nueva proteína que se va sintetizar).
Las principales características del código genético son:
a) El código genético está degenerado. Combinando las cuatro bases nitrogenadas del ARNm en tripletes o codones, existen hasta 64 posibles codones distintos. En cambio, sólo existen 20 aminoácidos, por lo un mismo aminoácido puede estar codificado por más de un codón. Por ejemplo, los codones GAA y GAG especifican el ácido glutámico (redundancia), pero ninguno especifica otro aminoácido (no existe ambigüedad). Todos aminoácidos, salvo la metionina y el triptófano, están codificados por más de un codón (codones sinónimos).
Esto puede ser una ventaja, ya que si por error se cambia un nucleótido, puede ser que no codifique otro aminoácido distinto y se genere otra proteína.
b) Algunos codones, como UAA, UAG, o UGA no codifican para ningún aminoácido, pero indican una señal de terminación del proceso de traducción.
c) El codón AUG codifica para el aminoácido metionina y también como señal de iniciación. Así, el primer AUG en un ARNm es la zona que señala el lugar donde comienza la traducción de proteínas.
d) El código genético es universal, ya que está compartido por todos los seres vivos conocidos. Así, el mismo codón codifica para el mismo aminoácidos en todos los seres vivos conocidos. Por ejemplo, el codón UUU codifica el aminoácido fenilalanina tanto en procariotas como en eucariotas. Esto puede probar que todos los seres vivos conocidos tienen un mismo origen. Aunque se usa el término "universal" se refiere únicamente a la vida terrestre, ya que no se ha comprobado la existencia de vida en otro planeta.
e) No presenta imperfección/ambigüedad: ningún codón puede codificar más de un aminoácido, ya que si lo hiciera, sería un gran problema que un mismo gen codificase proteínas diferentes.
f) Es un código sin solapamientos. Los tripletes están dispuestos de manera lineal y continua, sin que entre ellos existan espacios y sin que compartan ninguna base nitrogenada. Su lectura se hace en un solo sentido (5´→3´), desde el codón de iniciación, que indica el comienzo de la proteína, hasta el codón de parada que indica su final. Sin embargo, un mismo ARNm puede tener varios codones de iniciación, lo que significa que se podrían sintetizar varios polipéptidos distintos a partir del mismo ARNm (esto es típico de procariotas).
3.5. Traducción del ARNm a proteínas
El proceso de traducción consiste en la transformación de la información aportada por la secuencia nucleótidos del ARNm en una secuencia de aminoácidos.
La traducción es similar en procariotas y en eucariotas, pero hay alguna diferencia. Por ejemplo, el ARNm de los procariotas no necesita maduración, por lo que según se sintetiza, es leído por los ribosomas para traducir su información a aminoácidos de una proteína.
En cambio, en las células eucariotas, el ARNm transcrito primario se sintetiza en el núcleo y tiene que seguir un proceso de maduración antes de convertirse en ARN funcional que atraviese los poros de la membrana nuclear hacia el citoplasma. Después, en el retículo endoplasmático rugoso o en el citosol, los ribosomas traducirán su información a proteínas.
Se distinguen las siguientes etapas consecutivas en la biosíntesis de las proteínas: activación de los aminoácidos, iniciación de la traducción, elongación de la cadena polipeptídica y terminación de la traducción.
A) ACTIVACIÓN DE LOS AMINOÁCIDOS O FORMACIÓN DEL COMPLEJO AMINOÁCIDO-ARNt
Esta fase previa tiene lugar en el citoplasma y no en los ribosomas.
Antes de comenzar la síntesis de proteínas es necesario que el aminoácido se active, uniéndose al triplete CCA del ARNt. Las enzimas aminoacil-ARNt sintetasas son específicas para cada aminoácido, y se encargan de realizar esta unión, pero es necesaria la energía proporcionada por la hidrólisis de ATP. Existen 20 aminoacil-ARNt-sintetasa, una para cada aminoácido. Estas enzimas son muy específicas, pues han de unir cada aminoácido al ARNt correspondiente.
El aminoácido se une por su extremo carboxilo (-COOH) al extremo 3' del ARNt (concretamente, al grupo hidroxilo (-OH) del carbono 3' del último nucleótido, que siempre lleva adenina), y pasa a llamarse aminoacil-ARNt.
Aminoácido + ATP + ARNt ↔ aminoacil-ARNt + AMP + PPi
Como recordarás, las moléculas de ARNt tienen cuatro “brazos”: D, T, aceptor de aminoácidos y anticodón.
En el brazo aceptor de aminoácidos se sitúan los extremos 3' y 5' de la cadena:
a) En el extremo 3' siempre aparece el triplete CCA, al que se une el aminoácido.
b) El extremo 5' siempre termina con un nucleótido de guanina.
El brazo anticodón contiene un triplete de bases nitrogenadas específico para cada tipo de ARNt. Tiene la función de unirse al correspondiente codón complementario del ARNm.
Las moléculas de ARNt son las intermediarias entre la secuencia de nucleótidos del ARNm y la secuencia de aminoácidos, ya que, además de aportar el aminoácido que llevan unido en su extremo 3', se encargan de reconocer el codón de ARN complementario a su anticodón.
B) INICIACIÓN DE LA TRADUCCIÓN
El ARNm se une, por su extremo 5´, a la subunidad menor de los ribosomas gracias a una secuencia inicial llamada región líder, que no se traduce, en la que hay unos 10 nucleótidos complementarios con el ARN ribosómico. El ARNm se desplaza hasta que llegar al codón AUG, que codifica el aminoácido metionina y es el triplete que actúa como señal de iniciación.
Como vemos, el primer triplete que se traduce es el codón de iniciación AUG y, por tanto, el anticodón del primer ARNt tiene que ser UAC, su complementario. Y la metionina, será siempre el primer aminoácido de la cadena peptítica, aunque se suele eliminar al finalizar la traducción.
La subunidad menor del ribosoma, junto con el ARNm y el primer aminoacil-ARNt forman el complejo de iniciación, al que después se unirá la subunidad mayor del ribosoma.
Entonces se les une el complejo formado por el aminoacil-ARNt. La unión se realiza entre el codón del ARNm y el anticodón del ARNt que lleva el aminoácido. Por último, se une la subunidad mayor a la menor completándose el ribosoma.
Al finalizar esta etapa, la subunidad mayor de ribosoma se acopla al complejo de iniciación, formando el ribosoma completo. Este complejo ribosomal o complejo activo tiene dos lugares de unión:
a) El sitio peptidil o sitio P, que ocupa el primer aminoacil-ARNt, el ARNt-metionina.
b) El sitio aminoacil o sitio A, que está libre y preparado para recibir al segundo ARNt con otro aminoácido.
Todos estos procesos precisan gasto de GTP.
C) ELONGACIÓN DE LA TRADUCCIÓN
La elongación consiste en la adición de aminoácidos al extremo carboxilo de la cadena polipeptídica. Comienza cuando otro aminoacil-ARNt, con un anticodón complementario al codón del ARNm que está a continuación del codón iniciación, AUG, ocupa el sitio A (aminoacil) del ribosoma que estaba libre.
El grupo carboxilo del primer aminoácido se une por un enlace peptídico con el grupo amino del segundo aminoácido, catalizado por la enzima peptidil-transferasa.
El sitio P (peptidil) estará entonces ocupado por un ARNt sin aminoácido, ya que se habrá unido al que transportaba el ARNt que ahora ocupa el sitio A.
Después se produce la translocación ribosomal, desplazándose el ribosoma tres nucleótidos en sentido 5’→3’, liberando el ARNt que antes tenía la metionina y que ocupaba el sitio P. El ARNt con el dipéptido recién formado que ocupaba el sitio A, pasará a ocupar el sitio P, quedando vacío el sitio A.
Este sitio A volverá a ser ocupado por otro aminoacil-ARNt con un anticodón complementario al codón a traducir, y así sucesivamente. Para todos estos procesos se necesita energía que obtiene del GTP.
Así se forma la cadena peptídica, uniendo los sucesivos aminoácidos que van llegando al ribosoma transportados por los ARNt correspondientes.
D) TERMINACIÓN DE LA TRADUCCIÓN
La elongación continúa hasta que el ribosoma llega a uno de los codones de terminación (UAA, UAG y UGA), que es la señal que indica que ha terminado la traducción. No hay ningún ARNt que tenga un anticodón complementario a estos codones de terminación, por lo que el sitio A no será ocupado por ningún aminoacil-ARNt y terminará la cadena peptídica.
La velocidad de la síntesis de proteínas es muy alta, ya que pueden llegar a unirse hasta 1400 aminoácidos por minuto.
Normalmente, las cadenas de ARNm son traducidas por más de un ribosoma a la vez, y forman los polirribosomas o polisomas, lo que permite que la traducción sea mucho más eficiente y rápida.
En los procariotas, como no existe membrana nuclear, la transcripción y traducción se producen simultáneamente y el ARNm comienza a traducirse antes de estar completamente transcrito.
4. Mutaciones
El ADN como material hereditario se transmite con gran fidelidad a las generaciones siguientes. Aunque las ADN polimerasas sintetizan ADN con una tasa de error muy pequeña y tienen mecanismos para reparar sus errores, alguna vez se producen alteraciones. Estos cambios producidos en la replicación del ADN son las mutaciones.
Hugo de Vries, además de ser uno de los que redescubrió las leyes de Mendel en 1900, fue el primero que utilizó el término mutación al estudiar los cambios bruscos que aparecían en la descendencia de unas plantas de Oenothera lamarkiana. Definió mutación como “cualquier cambio heredable en el material hereditario que no se puede explicar mediante segregación o recombinación”.
Morgan observó que en los cultivos de Drosophila, incluso en aquellos que habían demostrado su condición de razas puras durante muchas generaciones, aparecían con cierta frecuencia variantes morfológicas hereditarias cuya presencia sólo resultaba explicable si se admitía que los genes responsables de tales variantes habían sufrido una mutación.
En la terminología genética es frecuente el uso de las expresiones alelo normal y alelo mutante pare referirse respectivamente al alelo más frecuente y al menos frecuente en una población. Esta terminología puede resultar útil en casos en los que, como ocurre con las variantes morfológicas estudiadas en Drosophila, el fenotipo determinado por el alelo mutante es extremadamente raro en las poblaciones naturales. Sin embargo, en otros muchos casos, como por ejemplo el grupo sanguíneo o el color del pelo en la especie humana, no es posible, y además carece de interés, determinar cual es el alelo normal y cual el mutante.
Esta definición cambio cuando se conoció que el material hereditario es el ADN y la estructura de doble hélice del ADN (Watson y Crick, 1953). Se definió mutación como cualquier cambio en la secuencia de nucleótidos del ADN.
Los estudios experimentales realizados acerca de la mutación durante varias décadas demuestran que es un fenómeno aleatorio. Cada gen lleva asociada una determinada probabilidad, que es constante de generación en generación, de sufrir una mutación que dé lugar a un determinado alelo, pero es impredecible el momento en que esta mutación va a tener lugar.
Los cambios en el ADN también implican cambios en la secuencia de aminoácidos que constituyen la proteína correspondiente, por lo que las mutaciones pueden afectar a la supervivencia del organismo.
4.1. Tipos de mutaciones
Las mutaciones se pueden clasificar según varios criterios:
A) SEGÚN EL TIPO DE CÉLULAS AFECTADAS:
· Somáticas: No se heredan, ya que sólo se transmiten a células que se originan por mitosis. Son las mayoritarias. No afectan a la evolución.
· Germinales: Afectan a los gametos o a las células que los produce, y se transmiten a la descendencia. Sobre ellas actúa la selección natural.
B) SEGÚN SU CAUSA:
· Naturales o espontáneas.
· Inducidas por agentes mutágenos.
C) SEGÚN SUS EFECTOS:
· Beneficiosas. Algunas mutaciones (muy pocas) mejoran el funcionamiento de la proteína que codifican.
· Perjudiciales. Distinguimos las mutaciones letales (producen la muerte de al menos el 90% de los que las sufren), subletales (mueren menos del 10% de los que las padecen) o patológicas (producen alguna enfermedad).
· Neutras. Son silenciosas, ni benefician ni perjudican.
Aunque la mutación se transmita a la descendencia, a veces puede no manifestarse. Si es un carácter dominante sí se puede apreciar fácilmente, pero si es recesivo, es difícil detectarlo, ya que sólo se manifiesta si el individuo es homocigótico recesivo.
La mutación, junto con la recombinación meiótica, es la fuente de variabilidad genética que permite la evolución.
Aunque sólo se producen aproximadamente 10-5 mutaciones por gen y generación, en un organismo diploide como los humanos, con unos 105 genes, las mutaciones que tiene cada individuo al nacer son de 10-5 x 105 x 2 = 2 mutaciones, lo que es una cantidad considerable.
4.1.1 Mutaciones génicas o puntuales
Las mutaciones génicas son lo que hoy en día se consideran mutaciones en sentido estricto. Consisten en cambios químicos del ADN que afectan tanto a los genes estructurales como a los reguladores, y pueden provocar cambios en un par de bases (microlesiones) o en un segmento génico (macrolesiones).
Aparecen básicamente por dos causas:
a) Errores no corregidos durante la replicación del ADN.
b) Acción de determinados agentes físicos o químicos (mutágenos).
Podemos distinguir varios tipos de mutaciones génicas:
A) Mutaciones por sustitución de una base por otra distinta; dentro de estas tenemos:
· Transiciones; una base púrica es sustituida por otra base púrica o una pirimidínica por otra pirimidínica.
· Transversiones; una base púrica es sustituida por otra pirimidínica (o viceversa).
B) Mutaciones por pérdida o inserción de bases; estas mutaciones son mucho más graves que las anteriores porque no afectan a un solo triplete de bases. A partir del punto de deleción o adición, TODOS los tripletes de bases estarán cambiados y, por lo tanto, el mensaje codificado será totalmente diferente.
Se suelen producir por un emparejamiento anómalo durante la replicación entre la hebra molde y la que se está sintetizando o cuando ciertos compuestos se intercalan en la cadena polinucleótida.
4.1.2 Mutaciones cromosómicas
Este tipo de mutaciones afecta a la estructura de los cromosoma y, por lo tanto, es posible detectarla al microscopio. La secuencia de nucleótidos de los genes no está alterada, pero hay cambios en el número de estos o en su disposición en los cromosomas. Según se vean afectados el orden o número de los genes en los cromosomas, se diferencian dos tipos de mutaciones cromosómicas:
A) Alteraciones en el orden de los genes; no perjudican al individuo que las sufre pero causan la producción de gametos anormales que darán lugar a una descendencia con déficit o exceso de genes. Podemos distinguir dos tipos:
· Inversiones; la disposición de los genes de un fragmento cromosómico está invertida.
· Translocaciones; un fragmento cromosómico cambia de posición, trasladándose a otro lugar del mismo cromosoma, a su homólogo o a otro cromosoma cualquiera. Si la translocación se produce de un cromosoma a otro y de este al primero se denomina recíproca; si el segmento simplemente pasa a situarse en otro cromosoma se llama transposición.
B) Alteraciones por la existencia de un número incorrecto de genes; tienen lugar por un fallo en el apareamiento meiótico que puede producir un sobrecruzamiento erróneo, quedando un cromosoma con un fragmento extra y el otro con un déficit. También pueden resultar de inversiones o translocaciones en los parentales. Al contrario que los anteriores sí tienen efectos fenotípicos y en la mayoría de los casos deletéreos (mortales). Los gametos obtenidos originarán, tras la fecundación, diversas anomalías, como las siguientes:
· Deficiencias y deleciones; consisten en la pérdida de un fragmento del cromosoma y, en consecuencia, de algunos genes, ya sea en el extremo (deficiencia) o en otro lugar (deleción).
· Duplicaciones; un segmento de un cromosoma se encuentra repetido, por lo que existe un exceso de los genes correspondientes. En términos evolutivos, las duplicaciones poseen una importancia extraordinaria, ya que el aumento del número de genes puede determinar la aparición de nuevas variantes génicas en mutaciones posteriores.
Un ejemplo clásico de mutación cromosómica estructural en humanos es el síndrome “cri du chat” (grito de gato) provocado por una deleción particular en el cromosoma 5 y que se caracteriza por microcefalia, retraso mental profundo y detención del crecimiento. El nombre viene derivado del llanto de estos niños que recuerda al maullido de un gato.
Como ya dijimos anteriormente, estas mutaciones son visibles al microscopio así que pueden detectarse mediante el bandeo cromosómico. Cada cromosoma tiene un patrón típico de bandas transversales así que comparando el patron de bandas del cromosoma a estudiar con el patrón conocido de dicho cromosoma podemos detectar si hay modificaciones.
Otra forma de observar estas mutaciones es cuando se produce el emparejamiento de los cromosomas homólogos en la profase I de la meiosis.
Estas mutaciones son MUY importantes a nivel evolutivo (excepto la deleción que carece de ningún efecto evolutivo). Un caso paradigmático es el de los humanos cuyo cromosoma 2 procede de la fusión de dos cromosomas de un homínido antropomorfo primitivo. Algunos genes que provocan la hemofilia también se sabe que se han adquirido por duplicaciones ocurridas en el transcurso de la evolución.
4.1.3 Mutaciones genómicas o numéricas
Las mutaciones genómicas o numéricas consisten en la alteración del número de cromosomas de una especia, ya sea por exceso o por defecto, por lo que se pueden detectar fácilmente al estudiar el cariotipo (conjunto de rasgos característicos de los cromosomas de una especie concreta) de un individuo.
Producen siempre alteraciones graves, pues cada cromosoma es portador de un elevado número de genes. Las más tolerables, lógicamente, son las que afectan a cromosomas pequeños. Se distinguen dos tipos: euploidías y aneuploidías.
A) Euploidías; se trata de una alteración en el número de juegos cromosómicos. Se denomina juego cromosómico al conjunto constituido por un cromosoma de cada tipo, por lo que los individuos diploides normales tienen en sus células somáticas dos de ellos. Podemos distinguir dos tipos:
· Monoploidías; únicamente existe un juego cromosómico completo (n cromosomas).
· Poliploidías; hay más de dos juegos cromosómicos. Pueden ser triplloidías (3n), tetraploidías (4n), hexaploidías (6n), ...
Estas mutaciones ocasionan un aumento del tamaño celular que puede ir acompañado de un aumento del tamaño corporal, lo cuál sucede con frecuencia en vegetales. Por esta razón muchas plantas de cultivo son poliploides (platanos → 3n, patatas → 4n, trigo → 6n). Los organismos poliploides pueden ser autoploides, cuando todas las dotaciones cromosómicas pertenecen a la misma especie, o alopoliploides, cuando proceden de la hibridación de especies distintas.
En los animales poliploides el aumento celular no se traduce en un aumento de tamaño del animal y, al tener menos células, son menos funcionales y menos viables.
B) Aneuploidías; no existe alteración del número de juegos cromosómicos completos. Solamente falta o sobra algún cromosoma individual.
Podemos distinguir cuatro tipos:
· Nulisomías (2n-2); falta una pareja cromosómica y es siempre letal.
· Monosomías (2n-1); falta un cromosoma de una determinada pareja. En humanos solo hay una monosomía viable: El síndrome de Turner que presenta un solo cromosoma X (X0)
· Trisomías (2n+1); un cromosoma se encuentra triplicado. Es el caso con más afectados en humanos. Hay tres trisomías que afectan a autosomas [síndrome de Patau (13), el síndrome de Edwards (18) y el síndrome de Down (21)] y otras que afectan a los heterosomas o cromosomas sexuales [síndrome de Klinefelter (XXY), el síndrome del "superhombre" (XYY) o el síndrome triple X (XXX).
· Tetrasomías (2n+2); existen cuatro ejemplares de un determinado cromosoma.
Las aneuploidías se originan por la fusión de un gameto normal (n cromosomas) con otro anormal (n-1, n+1, n+2). Las más tolerables son aquellas que afectan a cromosomas pequeños y a los cromosomas sexuales.
4.2. Agentes mutagénicos
Desde que Muller (1927) y Stadler (1928) comprobaron que la aplicación de rayos X sobre las moscas de la fruta y el centeno, respectivamente, inducía la aparición de mutaciones, se han descubierto muchos agentes mutagénicos, que se pueden clasificar en tres grupos: físicos, químicos y biológicos.
4.2.1. Agentes mutagénicos físicos
Las subidas intensas y rápidas de temperatura pueden producir mutaciones pero los agentes mutagénicos físicos típicos son las radiaciones. Dentro de estas distinguimos entre:
A) Radiaciones ionizantes; son radiaciones con una longitud de onda muy corta y, por lo tanto, muy energéticas, que provocan la ionización de los átomos de las sustancias que atraviesan. Entre estas radiaciones se encuentran los rayos X y γ, así como las partículas α y β y los neutrones emitidos en los procesos radiactivos.
Los efectos de las radiaciones ionizantes sobre los seres vivos son de tres tipos:
· Fisiológicos; pueden producir cambios enzimáticos que se traducen en alteraciones metabólicas.
· Citogenéticos; comportan alteraciones en la estructura del cromosoma (deleciones, translocaciones, ….).
· Genéticos; las ionizaciones directas del ADN o la formación de radicales libres muy reactivos originan cambios químicos en el ADN que se traducen en mutaciones génicas (rotura de enlaces nucleotídicos, rotura y pérdida de bases nitrogenadas, aparición de formas tautoméricas, …).
B) Radiaciones no ionizantes; básicamente se refiere a la radiación ultravioleta (UV). No tiene tanta energía como para producir ionizaciones pero sí provoca que los electrones pasen a niveles energéticos más altos lo cuál puede originar tautómeros y dímeros de timina.
4.2.2. Agentes mutagénicos químicos
Muchas sustancias químicas tienen acción mutagénica (hidrocarburos policíclicos, aminas aromáticas, agentes alquilantes, colorantes industriales, pesticidas, …). A diferencia de lo que ocurre con las radiaciones, sus efectos suelen ser más retardados en el tiempo. Los principales cambios que pueden provocar son:
a) Modificaciones de bases nitrogenadas; comprenden las reacciones de desaminación, alquilación e hidroxilación, que provocan emparejamientos erróneos. Así, por ejemplo, la desaminación de la adenina da lugar a la hipoxantina, que se aparea con la citosina en lugar de con la timina; la guanina se transforma, mediante alquilación, en 6-O-metilguanina, que se aparea con la timina en lugar de con la citosina.
b) Sustituciones de bases; están causadas por análogos de bases nitrogenadas que provocan un emparejamiento erróneo durante la replicación, al cambiar una base por otra. Entre ellos se encuentran la 2-aminopurina y el 5-bromouracilo.
c) Introducción de ciertas moléculas en la cadena polinucleotídica del ADN; estas inserciones provocan la aparición de un exceso de nucleótidos en la hebra de nueva formación durante la replicación. A partir de ese punto, los tripletes de bases se alteran y el mensaje resultante cambia.
4.2.3. Agentes mutagénicos biológicos
Algunos agentes biológicos aumentan la frecuencia de las mutaciones génicas. Podemos destacar ciertos virus que pueden producir cambios en la expresión de algunos genes (retrovirus, adenovirus, virus de la hepatitis B, …) y los transposones.
Se han encontrado transposones en todo tipo de organismos y pueden causar mutaciones importantes al causar una activación o inactivación génica no deseada al insertarse en los genes estructurales o en los reguladores. También se cree que los virus mutagénicos podrían realizar su acción al llevar en su genoma transposones tomados de una célula infectada anteriormente.
El cáncer es causado por un proceso de división celular sin control que provoca la multiplicación rápida y desorganizada de las células. Esta multiplicación conduce a la destrucción del tejido afectado e, incluso, a la invasión de otros órganos (metástasis).
En el desencadenamiento de un proceso cancerígeno intervienen múltiples factores pero está claro que hay una fuerte relación entre determinados cambios en el ADN y la aparición de células cancerosas. También muchos agentes mutagénicos son agentes cancerígenos (radiaciones, virus, productos químicos, …).
Aunque todavía queda mucho por aprender del proceso de malignización de una célula se han producido grandes avances en su estudio en los últimos años. De forma muy resumida el principal problema es que defectos en determinados genes que regulan la división celular provocan que esta se vuelva caótica y se divida de forma descontrolada. En este proceso intervienen dos tipos de genes:
a) Oncogenes (onkos=tumor, genos=origen); provocan un aumento de las señales que estimulan la división celular, sin que estén presentes los estímulos normales para ello. De esta forma, se promueve la proliferación continua de las células. Hasta la fecha se han descubierto más de cincuenta oncogenes en varias especies, entre ellas la humana.
Actualmente se cree que los oncogenes proceden de otros genes, denominados protooncogenes, que codifican proteínas implicadas en determinadas etapas de la división celular (factores de transcripción, factores extracelulares estimulantes o receptores de membrana para estos últimos). La alteración de los protooncogenes por acción de agentes mutagénicos originaría los oncogenes activos. Existen indicios de que en el proceso de malignización de una célula intervienen otros factores que favorecen la expresión de los oncogenes.
b) Genes supresores de tumores; la mutación de estos genes, que codifican proteínas inhibidoras de la división celular, estimula el aumento del ritmo reproductor de las células.
Los agentes mutagénicos podrían actuar en ambos sentidos y es probable que, para que se desarrolle un tumor, sean necesarias varias mutaciones en diversos genes.
Por otra parte, la mutación de los genes implicados en la corrección de errores del ADN evitaría la reparación de estos tras la actuación del agente mutagénico, en las primeras fases del proceso, y contribuiría notablemente al desarrollo definitivo del tumor.
5. Biotecnología
La biotecnología es la aplicación de la tecnología a los procesos biológicos. Utiliza organismos vivos, o sus derivados, para la obtención de productos o procesos.
Los seres humanos han usado la biotecnología desde la prehistoria para fabricar productos como el pan, el queso, la cerveza, …. También para mejorar a sus productos agrícolas y ganaderos.
Actualmente, el gran avance del conocimiento científico, nos permite ampliar los campos y el alcance de utilización de la biotecnología. Por eso se distinguen normalmente dos tipos: la biotecnología tradicional y la biotecnología actual.
5.1 Biotecnología tradicional
La biotecnología tradicional se basa, principalmente, en la utilización de microorganismos para obtener productos útiles para el ser humano. Podemos destacar tres áreas en las que se han aplicado estas técnicas:
a) Usos alimentarios
· Fabricación del pan gracias a las fermentaciones alcohólicas llevadas a cabo por la levadura Saccharomyces cerevisiae.
· Fabricación del queso y el resto de derivados lácteos mediante la fermentación láctica que realizan algunas bacterias.
· Producción de bebidas alcohólicas a partir de las fermentaciones llevadas a cabo por la levadura Saccharomyces cerevisiae.
b) Usos sanitarios
· Fabricación de vacunas utilizando microorganismos atenuados o fragmentos de los mismos.
· Elaboración de antibióticos producidos por ciertos mohos.
c) Usos medioambientales
· Uso de bacterias para la descomposición de la materia orgánica de los residuos sólidos urbanos mediante fermentaciones.
· Eliminación de la materia orgánica en las depuradoras mediante acciones realizadas por bacterias.
· Fabricación bacteriana de bioplásticos, que son biodegradables.
· Eliminación de los vertidos de petróleo mediante su degradación por bacterias especializadas.
5.2 Biotecnología actual. Ingeniería genética
La biotecnología actual nace a finales del siglo XX y está basada en los descubrimientos y logros de la ingeniería genética.
La ingeniería genética es el conjunto de técnicas para manipular el material genético de un organismo para transferirlo a otro y que se exprese en él.
5.2.1 Técnicas de ingeniería genética
Las técnicas de trabajo más empleadas en ingeniería genética son la obtención de ADN recombinante, la utilización de vectores y la técnica de reacción en cadena de la polimerasa (PCR).
5.2.1.1 ADN recombinante
El ADN recombinante es una molécula de ADN artificial obtenida a partir del gen de un organismo y de un vector. La técnica para obtener ADN recombinante se utiliza mucho en investigación y consta de cuatro fases básicas:
a) Se localiza el gen que se va a manipular y se analiza su secuencia de nucleótidos.
b) Se aísla el gen utilizando unas enzimas (enzimas de restricción) que cortan el ADN en zonas específicas.
c) El gen se une a un vector (ADN de una bacteria o de un virus), que lo transportará. La unión gen+vector es el ADN recombinante.
d) El ADN recombinante se introduce en la célula que expresará la proteína. Esta se dividirá produciendo numerosas células hijas que producirán esa proteína.
Mediante esta técnica se consigue generar muchas copias a partir de un fragmento de ADN en un periodo corto de tiempo . Fue desarrollada en 1983 por Kary Mullis y permite clonar fragmentos de ADN sin usar ninguna célula, directamente en un tubo de ensayo.
Para esta reacción es necesario:
· La enzima ADN polimerasa, resistente al calor (se obtiene de alguna bacteria termorresistente).
· Un cebador. Un pequeño fragmento de ARN de unos 20 nucleótidos, necesario para que pueda actuar la ADN polimerasa.
· Una fuente de calor.
· Nucléotidos de ADN.
El procedimiento sigue los siguientes pasos:
a) Se calienta la molécula de ADN que se quiere copiar a una temperatura superior a 90 ºC, para que se desnaturalice y pierda su estructura, separándose las dos cadenas que forman la doble hélice (se rompen los puentes de hidrógeno que unen las bases nitrogenadas de ambas cadenas).
b) Cada una de las dos cadenas sirve de molde para una nueva cadena complementaria.
c) El cebador, un fragmento de ARN, permite que la enzima ADN polimerasa pueda continuar añadiendo desoxirribonucleótidos (nucleótidos de ADN) hasta formar toda la cadena complementaria.
d) Después, las cadenas recién formadas se vuelven a separar por efecto del calor, comenzando un nuevo ciclo.
Así, en poco tiempo se pueden conseguir muchísimas copias del fragmento de ADN original.
La clonación es una técnica que permite obtener una copia idéntica de una molécula, célula u organismo ya desarrollado, a partir de su ADN. Dos clones son individuos genéticamente idénticos.
La ingeniería genética permite desarrollar tres tipos de clonación:
a) Clonación molecular; permite hacer copias de ADN mediante unas células llamadas células anfitrionas, aunque hemos visto en el apartado anterior otra técnica, la PCR o reacción en cadena de la polimerasa que es mucho más rápida.
b) Clonación de células; se pueden obtener células genéticamente idénticas que se pueden utilizar para reparar tejidos enfermos sin que se produzca rechazo.
c) Clonación de organismos completos; se obtienen individuos genéticamente idénticos.
La clonación, obtener copias idénticas, es un proceso natural y frecuente en los organismos de reproducción asexual. En cambio, con la reproducción sexual, los individuos generados son distintos entre ellos y a sus progenitores. La única excepción es la de los gemelos monocigóticos, que provienen del mismo óvulo y mismo espermatozoide, que son genéticamente iguales.
Existen dos tipos de clonación en animales: la clonación reproductiva y la clonación terapéutica.
Básicamente, la clonación reproductiva consiste en obtener un individuo idéntico a otro extrayendo el núcleo (2n) de una célula somática del individuo original e introduciéndolo en el óvulo de otro individuo hembra de esa misma especie al que se le ha extraído el núcleo (n). Así, el óvulo tiene un núcleo (2n) se comporta como si hubiera sido fecundado y se divide hasta formar un embrión. Este embrión se implanta en el útero de otra hembra. El embrión dará lugar a un animal idéntico al donador del núcleo (2n) que se introdujo en el óvulo, ya que que tendrá su mismo ADN. De este modo fue como nació en 1996 el primer mamífero clonado, la oveja Dolly.
La clonación de animales puede servir para llegar a recuperar especies en peligro de extinción o incluso ya extinguidas, mejorar las razas de ganado o producir animales que generen proteínas humanas o medicamentos.
No está permitida legalmente la clonación de humanos, pero sí que se puede utilizar la clonación terapéutica para curar algunas enfermedades o en trasplantes de órganos. Para hacerlo, es necesario obtener células madre, aunque también hay polémica sobre si es legítimo obtenerlas a partir de embriones que no se utilizan en la reproducción asistida o si sólo se deben extraer de tejidos y órganos adultos.
Las célula madre son células pluripotentes, indiferenciadas, que no tienen aún ninguna función específica pero que pueden convertirse en diferentes células del organismo. Cuando se divide una célula madre, las células hijas pueden seguir siendo células madre o transformarse en otro tipo de célula más especializada, como una neurona.
Con células madre se pueden reparar tejidos dañados por infartos, quemaduras, fracturas,..., y se está investigando para su aplicación en algunas enfermedades como la diabetes, Alzheimer, o leucemia.
5.2.3. Proyecto Genoma Humano
El Proyecto Genoma Humano fue un proyecto de investigación científica que tenía el objetivo fundamental de determinar la secuencia de pares de bases nitrogenadas del ADN e identificar y cartografiar los aproximadamente 25 000 genes del genoma humano desde un punto de vista físico y funcional. Se trataba de conocer la secuencia de nucleótidos de nuestro ADN y qué genes codifican las proteínas.
El proyecto comenzó en 1990, cuando se creó un Consorcio Público Internacional, formado por EEUU, Reino Unido, Japón, Francia, Alemania, China y otros países, con el objetivo de conseguir la secuencia completa del genoma humano. Paralelamente, otro proyecto similar fue impulsado por la empresa privada Celera Genomics, que pretendía hacer negocio con las patentes de los genes.
Finalmente, en 2001, se hicieron públicos de forma conjunta, la empresa pública y la privada, de la secuencia completa del genoma humano.
Algunas de las principales conclusiones obtenidas del Proyecto Genoma Humano son:
a) Tenemos unos 25 000 genes de tamaño variable, formados por unos 3000 millones de nucleótidos.
b) Compartimos más del 99,9 % con todas las demás personas, son idénticos. El 0,1 % es lo que hace que las personas seamos distintas a los demás.
c) Compartimos muchos genes con otras especies. por ejemplo, compartimos más del 90 % de los genes con los ratones y el 98 % con los chimpancés.
d) Sólo menos del 2 % del ADN codifica para proteínas, el resto no se sabe aún su función.
e) Cada gen codifica información para muchas proteínas, no solo una.
Los OMG u organismos transgénicos se denominan así porque se han obtenido introduciendo genes de otro ser vivo en su genoma. El objetivo de esto es transferirles propiedades que ellos no poseen o modificar las que presentan.
La incorporación de los genes se realiza directamente (con microagujas o cañones de genes) o a través de virus.
Los OGM tienen distintas aplicaciones como por ejemplo:
a) El uso de bacterias para la fabricación de productos terapéuticos, como la insulina o la hormona del crecimiento, o vacunas como la de la hepatitis B.
b) La utilización de virus necesarios en terapias génicas.
c) La modificación de animales con el fin de potenciar determinadas características deseables, por ejemplo un engorde más rápido.
d) El control de organismos generadores de plagas, como el mosquito que transmite la malaria.
e) La producción de plantas comestibles resistentes a plagas y herbicidas y la modificación de plantas de las que se obtienen alimentos mejorados, como el arroz dorado con provitamina A para evitar la ceguera en zonas muy pobres sin acceso a la carne.
Los OGM han sufrido fuertes campañas en contra argumentando riesgos sanitarios, medioambientales y económicos. Lo cierto es que son totalmente seguros para la salud y que su impacto medioambiental es menor al necesitar menos pesticidas, abonos, etc.
Un factor real que sí hay que tener en cuenta es que la mayoría han sido desarrollados y patentados por empresas privadas que obviamente cobran por su uso. Se deben potenciar las patentes públicas que garanticen la accesibilidad a los mismos por parte de todos los agricultores de todos los países.
El éxito que los científicos obtuvieron al clonar mamíferos generó especulaciones sobre la posibilidad de hacerlo con personas. Esto originó un problema ético nuevo, ya que por primera vez se planteaba la posibilidad de crear artificialmente un ser vivo de nuestra propia especie. En la actualidad, la ley prohíbe la clonación reproductiva en seres humanos.
La clonación terapéutica tiene importantes aplicaciones médicas al poder tratar ciertas enfermedades y reparar órganos dañados. Sin embargo, esta técnica no está exenta de polémica, ya que para obtener células madre se necesita crear embriones humanos que luego serán destruidos. Hoy día, muchos de los embriones utilizados se obtienen de donantes en tratamiento de fertilidad, pues la Ley de Investigación Biomédica de 2007 prohíbe la creación de embriones con fines de investigación.
El médico japonés Shinya Yamanaka fue galardonado en 2012 con el premio Nobel de medicina por estar considerado el padre de las llamadas células iPS, que poseen la capacidad de convertirse en cualquier tipo celular especializado. Desarrolló un método para la generación de células madre existentes en las células del cuerpo. Este método da lugar a la reversión de las células desde un estado adulto a un estado pluripotente.
Demostró cómo se pueden obtener las llamadas células madre pluripotentes a partir de células adultas. Las células pluripotentes tienen el potencial de diferenciarse en cualquier otra célula del organismo, por lo que se espera poder utilizarlas en un futuro próximo para regenerar órganos y tejidos dañados. El descubrimiento supuso una verdadera revolución, al dejar obsoleto el uso de su equivalente natural, las células madre embrionarias, cuya obtención plantea problemas éticos.
La Terapia génica es otro de los frentes abiertos por la biotecnología con más futuro. Podemos utilizar vectores como los virus para introducir genes terapéuticos que nos ayuden a prescindir de los fármacos.
Los
avances en la biotecnología y la ingeniería genética han abierto
el camino a muchas aplicaciones prácticas, como el diagnóstico,
eliminación, prevención y curación de enfermedades, la lucha
frente a la contaminación y la eliminación de residuos, la
obtención de nuevos combustibles, etc.
Frente a estos beneficios,
existe una lista de inconvenientes relacionados con los posibles
riesgos del uso de estas técnicas:
a) Riesgos éticos; la posibilidad de seleccionar las características de los hijos o de atentar contra determinados grupos de humanos, ….
b) Riesgos sociales; el aumento de la desigualdad por el acceso (o no) a estas técnicas según la renta y el país; la discriminación a personas en función de su genética de cara a obtener trabajos, seguros médicos, ...; ...
c) Riesgos medioambientales; la disminución de la biodiversidad al reducirse el número de variedades que se cultivan, …
d) Riesgos sanitarios; el introducir genes de otros organismos puede ocasionar problemas de alergias no detectadas, es posible que surjan enfermedades no conocidas, ...
e) Riesgos legales; la posibilidad de patentar productos biotecnológicos tiene multitud de implicaciones globales. Una de las principales controversias actuales es la posibilidad de patentar plantas y animales transgénicos o incluso secuencias del genoma humano. El Comité Europeo de Patentes prohíbe patentar genes humanos pero hay sectores que presionan para que esto cambie aludiendo a la necesidad de que las empresas ganen dinero y sigan investigando.
Para prevenir estos riesgos la UNESCO establece tres grandes recomendaciones: protección del ser humano, promoción del conocimiento y la investigación, solidaridad y cooperación internacional.










































































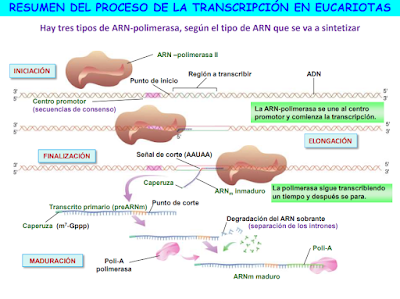









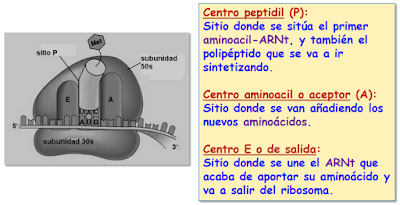
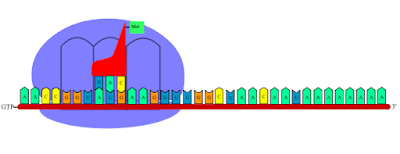


























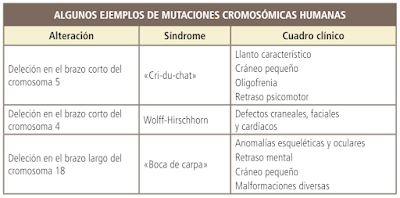


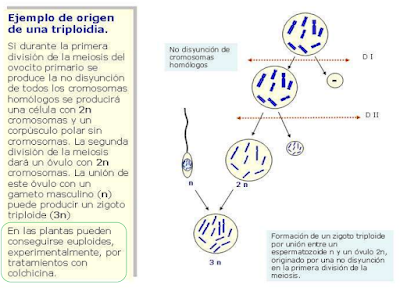


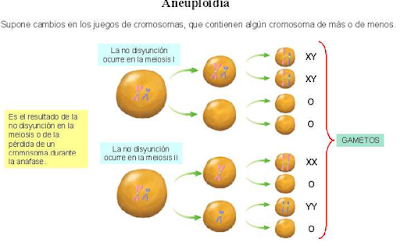





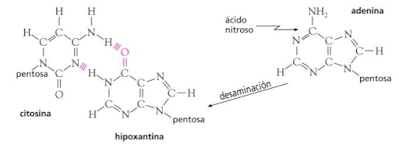
















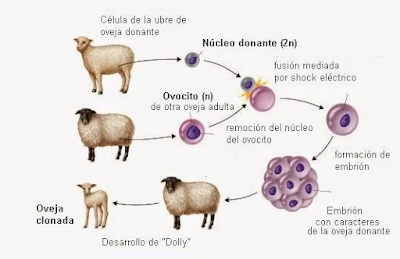









No hay comentarios:
Publicar un comentario